Loretta Sebastiani
Simulazione Esame di Stato.
Biologia DNA ricombinante.
Questa traccia viene proposta come simulazione in preparazione all’esame di stato per Biologia, microbiologia e tecnologie di controllo sanitario nella specializzazione di Biotecnologie sanitarie. La traccia è riferita nella sua parte obbligatoria alla tecnologia del DNA ricombinante e in maniera specifica ai vettori di espressione e ai meccanismi di trasformazione e trasfezione. Di questa parte è fornita anche la correzione. La parte facoltativa si articola in quattro domande che riguardano l’elettroforesi del DNA, l’analisi microbiologica degli alimenti, l’importanza del lievito Saccharomyces cerevisiae nelle produzioni biotecnologiche alimentari e, infine, i mutageni diretti con l’analisi del loro meccanismo d’azione e i relativi meccanismi di riparazione del DNA. Ulteriori dettagli sulla tecnologia del DNA ricombinante:
DNA RicombinanteESAME DI STATO DI ISTRUZIONE SECONDARIA SUPERIORE
Indirizzo: ITBS - CHIMICA, MATERIALI E BIOTECNOLOGIE
ARTICOLAZIONE BIOTECNOLOGIE SANITARIE
Tema di: BIOLOGIA, MICROBIOLOGIA, TECNOLOGIE DI CONTROLLO SANITARIO
Il candidato è tenuto a svolgere i quesiti della prima parte e due quesiti a sua scelta della seconda parte.
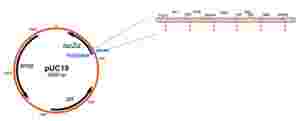
PRIMA PARTE
L’immagine sopra riportata si riferisce ad uno dei più comuni vettori ingegnerizzati utilizzati nella tecnologia del DNA ricombinante, creato da Joachim Messing. Il suo nome è l’acronimo di plasmid - University of California e 19 si riferisce al numero della serie.
Il candidato:
- dopo aver delineato le caratteristiche di un plasmide batterico, descriva nella sua interezza il vettore pUC19 rappresentato in figura mettendo in evidenza la successione delle aree e il motivo della loro presenza;
- descriva gli enzimi coinvolti dalla fase in cui si ottiene il gene di interesse a quella in cui il gene esogeno viene inserito nel vettore;
- spieghi le varie tecniche di trasformazione e trasfezione con particolare riguardo alla “competenza” delle cellule ospiti;
- evidenzi le diverse tecniche per amplificare in vivo e in vitro frammenti di DNA di interesse.
SECONDA PARTE
- Una delle tecniche di maggiore importanza nella tecnologia del DNA ricombinante è l’elettroforesi. Il candidato, illustrandone la metodica, ne sottolinei positività ed eventuali criticità. Analizzi poi la sua importanza in alcune tecniche di biologia molecolare.
- La qualità di un alimento ha un significato molto ampio perché al consumatore devono essere garantiti molti aspetti, dalle caratteristiche organolettiche a quelle microbiologiche. Il candidato, in base alla normativa che disciplina il controllo microbiologico degli alimenti, analizzi le modalità di campionatura dei prodotti alimentari, i microbi indicatori e le principali tecniche tradizionali e innovative utilizzate per la loro identificazione.
- Il candidato descriva i principali mutageni diretti in relazione al loro meccanismo d’azione, alle fonti di esposizione, al danno riportato dal DNA e ai relativi meccanismi di riparazione del DNA stesso.
- Saccharomyces cerevisiae è uno dei microrganismi più utilizzati nelle biotecnologie a tutti i livelli. Il candidato descriva i suoi possibili usi nella tecnologia del DNA ricombinante e nelle produzioni biotecnologiche alimentari.
SOLUZIONE DELLA PRIMA PARTE
- I plasmidi sono molecole di DNA circolari a doppio filamento presenti in quasi tutti i batteri. Sono molecole di DNA separate dal cromosoma batterico e in grado di autoreplicarsi in modo indipendente grazie alla loro personale origine di replicazione. In genere contengono geni che esprimono caratteristiche aggiuntive come il fattore della fertilità F o la resistenza agli antibiotici o la produzione di endotossine o ancora alcune vie metaboliche relative agli zuccheri.
L’immagine che apre la traccia raffigura il vettore ingegnerizzato pUC19 con i dettagli del sito multiplo di clonaggio (polylinker) comprensiva delle sequenze di riconoscimento su cui agiscono i vari enzimi di restrizione elencati.
Il vettore di 2,686 kpb si legge secondo il verso delle lancette dell’orologio e comprende varie aree di interesse oltre al polylinker (tra le paia di basi azotate 396 e 454).
lacZα, a monte del polylinker, è un gene che codifica per una parte dell’enzima β-galattosidasi e in questo caso funziona da marcatore, cioè consente di selezionare le cellule ospiti che hanno effettivamente incorporato il plasmide ingegnerizzato da quelle che invece non lo hanno incorporato.
ori è l’origine di replicazione a cui si lega il complesso di pre-replicazione che riconosce, apre ed inizia a copiare il DNA; la sua presenza è necessaria perché il plasmide avvii la sua duplicazione in maniera indipendente dalla cellula ospite consentendo quindi la clonazione in vivo e la gestione del numero di copie.
amp è un altro gene marcatore che porta le informazioni per la resistenza all’antibiotico ampicillina. - Gli enzimi utilizzati per ottenere il gene esogeno sono delle endonucleasi di restrizione, noti anche più semplicemente come enzimi di restrizione e scoperti intorno alla metà del secolo scorso studiando i batteri. I batteri hanno infatti la capacità di attaccare il DNA dei fagi frammentandolo; contemporaneamente proteggono il proprio acido nucleico attraverso la metilazione di alcune basi azotate. Nel giro di poco tempo questa specifica attività è stata trasferita ai DNA delle altre specie viventi per ottenerne i frammenti. Esistono attualmente centinaia di enzimi di restrizione che sono stati scoperti in varie specie batteriche. Il taglio del DNA avviene su una sequenza specifica e su ambedue i filamenti. La sequenza non è la stessa ma varia da enzima ad enzima. Ci sono però endonucleasi diverse che tagliano la stessa sequenza. Inoltre in molti casi la sequenza è palindroma (cioè si legge allo stesso modo in una direzione, su un filamento, e nella direzione opposta, sull’altro filamento).
Il taglio può essere simmetrico (blunt ends o estremità piatte) o sfalsato (sticky ends o estremità appiccicose).
La nomenclatura degli enzimi di restrizione prevede che le prime lettere si riferiscano alle iniziali della specie. Per esempio Eco richiama Escherichia coli. Una quarta lettera indica il ceppo. Infine si usa il numero romano che riguarda l’ordine della scoperta.
Quindi per ottenere il gene di interesse si sceglie uno specifico enzima di restrizione che non deve agire solo sul DNA “donatore” ma anche sul DNA del vettore.
Perché il gene esogeno possa essere inserito nel vettore deve poi intervenire l’enzima ligasi. - Si chiama trasformazione l’inserimento del vettore ingegnerizzato nella cellula ospite se si tratta di un batterio o di un lievito. Più opportuno il termine trasfezione quando la cellula ospite è superiore.
La trasformazione è un meccanismo di ricombinazione genica che avviene normalmente in natura tra batteri e che coinvolge anche i plasmidi. In pratica si tratta di un trasferimento genico orizzontale. Molto semplicemente i batteri sono in grado di incorporare molecole di DNA. Alcuni lo possono fare spontaneamente perché hanno recettori specifici; è il caso del Bacillus subtilis, una delle cellule più utilizzate in campo biotecnologico. Altri devono essere invece “resi competenti” aggiungendo per esempio alla reazione CaCl2.
La trasformazione può essere indotta anche attraverso elettroporazione, cioè sottoponendo le cellule ospiti e i vettori ingegnerizzati, posti in particolari cuvette nell’elettroporatore, a un brevissimo impulso elettrico. La scarica elettrica fa riarrangiare le molecole degli involucri esterni in modo che si formino dei pori utilizzabili per il passaggio del vettore. Lo svantaggio è che molte cellule batteriche possono morire in seguito a questo trattamento ma il loro numero è sempre inferiore a quello delle cellule che incorporano correttamente il vettore. La tecnica può essere utilizzata anche per le cellule vegetali.
Quando la cellula ospite ha l’ingombro della parete cellulare, come nel caso delle cellule vegetali, si può ricorrere alla formazione dei protoplasti. La loro parete cellulare viene digerita da opportuni enzimi e come protoplasti le cellule così modificate possono fondersi tra loro. Inoltre il protoplasto può far entrare direttamente il DNA se si aggiunge il polietilenglicole (PEG).
Da ultimo metodo bisogna ricordare il cannone genico. Il DNA viene adsorbito su particelle di oro o tungsteno microscopiche, con diametro di 1 micrometro. Queste particelle vengono letteralmente sparate attraverso uno strumento ad aria compressa. Alla velocità di 500 m/s raggiungono le cellule bersaglio, riuscendo a penetrare all’interno di esse. Le cellule presenti direttamente sulla traiettoria vengono sicuramente uccise ma quelle che si trovano in posizione concentrica si salvano e riescono ad incorporare il DNA. - La clonazione in vivo sfrutta l’inserimento del vettore all’interno della cellula ospite dove il vettore, che deve potersi autoduplicare in modo indipendente dalla cellula, può dare origine a molte copie di se stesso. Inoltre il vettore può essere separato facilmente dal DNA batterico.
La clonazione in vitro si basa invece su una tecnica che ha rivoluzionato la biologia: la PCR (Polymerase Chain Reaction - reazione a catena della polimerasi).
Maggiori dettagli sulla PCR:
...
